Introduction
1942 Beadle et Tatum : 1 gène → 1 π. |
 |
I. Colinéarité Gène – Protéine
Une protéine est une séquence linéaire non ramifiée d’acides aminés. L’ADN est une séquence linéaire non ramifiée de nucléotides. On pensait que le contrôle des protéines était séquentiel → colinéarité de séquence entre les nucléotides et les acides aminés. Cette relation a été mise en évidence par Yanofsky :

La Tryptophane Synthétase est constituée de 2 chaînes a et b. Il existe des mutants présentant une chaîne a modifiée. Pour chaque mutant, il a identifié sur la protéine l’acide aminé différent. Sur l’ADN, il a localisé les altérations. Ces modifications intervenaient dans le même ordre, et possédaient à peu près le même espacement. La colinéarité semblait parfaite : chaque acide aminé du polypeptide est codé par une région spécifique du gène.
II. Les ribosomes sont le siège de la synthèse des protéines
II.A. Mise en évidence 1953 – 1954 Zamecnick
| Injection d’acides aminés radioactifs, puis prélèvement de cellules de foie, puis broyage et centrifugation : les microsomes sont responsables de la synthèse des protéines. |  |
Autre expérience : broyat de cellules de foie de rat. |
 |
Les protéines radioactives sont sur les microsomes. On procède à une centrifugation. Le phénomène de la synthèse protéique ne nécessite pas l’intégrité cellulaire et peut se réaliser in vitro. Les différents constituants de la cellule continuent à fonctionner. |
 |
Les protéines radioactives sont localisées sur les microsomes. La centrifugation a permis d’isoler l’organite qui synthétise les protéines. Microsome = Vésicules + Ribosomes (ARN). Grâce aux détergents, on isole les ribosomes. Ce sont de petites particules fixées sur les microsomes qui sont à l’origine de la synthèse des protéines. Les grains de Pallade = ribosomes (riches en ARN). |
 |
II.B. Les ribosomes
II.B.1. Morphologie
Ce sont des particules globulaires sous forme libre chez les bactéries ou sous forme de polysomes. |
|
Chez les eucaryotes, on les trouve libres, sous forme de polysomes et sur le Réticulum Endoplasmique RE → RER = Ergastoplasme. On les trouve aussi dans les mitochondries et les chloroplastes. |
 |
II.B.2. Structure

Ils possèdent une structure externe poreuse qui contient de l’eau (70% du volume). Les protéines sont périphériques et les ARN centraux (détermination par les anticorps : si ils se fixent, les ARN sont à la périphérie). Détermination par montage : combinaisons. Les associations de molécules se font dans un ordre donné.1 molécule agit avec une autre, modifiant la structure de la protéine associée, fixe une troisième et ainsi de suite. Il s’agit d’un puzzle dont les pièces sont à géométrie variable (transitions allostériques).
II.B.3. Biogenèse
II.B.3.a. Chez les procaryotes
La synthèse des constituants s’opère au contact de l’ADN et la synthèse des protéines dans le hyaloplasme. Puis l’assemblage est spontané (pas de compartimentation cellulaire).
II.B.3.b. Chez les eucaryotes
- Synthèse des constituants
- Synthèse des ARN ribosomaux
Dans le nucléole |
Hors du nucléole 5S | dans le noyau.

La synthèse des ARN se fait dans la partie centrale fibrillaire. Chez l’homme, 10 chromosomes viennent dans la partie fibrillaire : ils contiennent les gènes des ARN ribosomaux (plusieurs milliers d’exemplaires répartis sur 10 chromosomes). Ces gènes identiques entre eux sont appelés gènes en tandem.
Au contact de la partie fibrillaire, il y création des ARN. Les gènes vont fabriquer les ARN en 45S qui vont être fragmentés.

La synthèse des protéines se fait dans le hyaloplasme. Puis auto-assemblage des sous unités dans la partie granulaire.

Compartimentation cellulaire et plusieurs gènes interviennent.
III. Les ARN sont des intermédiaires dans la synthèse des protéines
III.A. Indices laissant penser que les ARN sont des intermédiaires
- Les protéines peuvent être assemblées en l’absence d’ADN ® il existe donc un / des intermédiaires. Exemple : les hématies sans noyau synthétisent de l’hémoglobine pendant un certain temps.
- La synthèse des protéines se fait dans le hyaloplasme et l’ADN se trouve dans le noyau.
- Lorsque les cellules étaient riches en ARN, la synthèse protéique était importante.
- Les cellules n’effectuant pas de synthèse protéique importante sont pauvres en ARN.
- Lorsque la synthèse protéique augmente, elle est précédée d’une augmentation importante d’ARN.
- Expérience de Zamecnick : si on détruit l’ARN du surnageant, la synthèse des protéines ne se fait plus.
III.B. Mise en évidence de l’existence d’ARN messager 1960
III.B.1. Rejet de l’hypothèse 1 gène → 1 ribosome → 1 protéine
Un gène contrôle la synthèse d’une catégorie de ribosomes particulière spécialisée dans la synthèse d’une protéine donnée : spécificité de la machinerie. Arguments contre :
- Dans les bactéries
Dans l’ADN, le rapport (A+T)/(C+G) varie de 0,3 à 2,5 selon les espèces de bactéries. Dans les ribosomes, le rapport (A+U)/(C+G) varie de 1 à 1,5. D’une espèce à l’autre, il y a peu de différence pour les ribosomes alors qu’il existe de plus grandes différences sur l’information génétique. - Les ARN ribosomaux sont stables
La ½ vie est de plusieurs heures or les gènes peuvent s’exprimer rapidement en quelques minutes.
Exemple Colibacile :Glucose→ Lactose. En quelques minutes on voit apparaître les enzymes pour le lactose. Cette rapide apparition est en contradiction avec l’apparition lente et la longue durée de vie des ribosomes. Il existe d’autres ARN.
III.B.2. Hypothèse de l’existence d’un ARN messager
III.B.2.a. Mise en évidence de l’existence d’un ARNm 1960 Jacob – Meselson – Brenner
- 1 gène → 1 ribosome → 1 protéine
- 1 gène → 1 ARNm → 1 protéine sur 1 ribosome
Pour trancher entre ces 2 hypothèses, on a procédé de la façon suivante. E Coli cultivée sur milieu N15 produira des ribosomes N15. Ces bactéries sont replacées sur un milieu N14. On ajoute des bactériophages + U*, puis centrifugation au chlorure de césium.
- 1er résultat envisageable : si
ribosomes N15 appartenant à E Coli
ribosomes N14 fabriqués par gènes viraux
ET * sur ribosomes N14 alors l’hypothèse 1 est vérifiée.
- 2ème résultat envisageable : si
ribosomes N15 appartenant à E Coli
ribosomes N14 fabriqués par gènes viraux
ET U* sur ribosomes N15 (ARN du virus) alors l’hypothèse 2 est vérifiée.
Le résultat expérimental confirme la deuxième hypothèse. Il n’y a pas de ribosomes N14 : le virus est capable de fabriquer ses propres protéines sans passer par la synthèse de ribosomes. Il utilise ceux d’E Coli. Ribosome = machinerie universelle non spécifique. L’hypothèse 1, fausse, est définitivement rejetée. Il apparaît un nouvel ARN utilisé sur N15 = ARN messager.
III.B.2.b. Observation de l’ARNm – généralisation 1961 Watson F Gros
Si les ARNm n’avaient pas pu être observés, c’est parce que leur durée de vie est très courte. Technique de centrifugation en gradient de chlorure de césium + radioactivité.

4-5S trop petits pour coder pour l’ensemble d’une protéine.
ARNm trop peu nombreux pour être décelés ainsi. On fait appel à la radioactivité.
E Coli. Pulse de 30 s. U*.

ARN apparu très vite (pulse de 30’’). Même expérience en réalisant un pulse de 30’’, mais une chasse de 20’. La radioactivité est répartie de façon diffuse sur les ARN ribosomaux. La durée de vie des ARNm est très courte.

Les ARNm ont une densité de 14S. Il apparaissent rapidement (- de 30’’), disparaissent rapidement (- 20’). Les ARN 4-5S correspondent aux ARN de transfert.
ADN → ARNm → Protéine
Ribosome = machinerie universelle non spécifique.
III.C. Intervention des ARN de transfert
III.C.1. Hypothèse de l’existence d’un adaptateur
Problème de l’adaptateur : par quel mécanisme les acides aminés peuvent-ils être enchaînés dans l’ordre dicté par l’information génétique ? Il n’existe aucune affinité entre les acides aminés et les nucléotides. Les acides aminés ne viennent pas se positionner directement sur l’acide nucléique. Il y a donc un adaptateur. Une enzyme ? Faux.
Plus tard Crick démontre que l’adaptateur est en fait un petit ARN qui peut très facilement se fixer sur un ARN par complémentarité des bases.
III.C.2. Preuve que l’adaptateur existe et que c’est un ARN
III.C.2.a. Découverte de petits ARN 1957 Hoagland
Nécessaires aux synthèses in vitro, ils pouvaient fixer des acides aminés en présence d’ATP et d’enzymes. Ce sont le ARNt.
III.C.2.b. Preuve expérimentale de leur rôle 1962
Les ARNm étaient connus, et le code génétique aussi. Isolement de différents ARNt capables de fixer des acides aminés.

Si on met en présence ARNtCys + ATP + Cystéine + Enzyme spécifique (Cystényl ARNt synthétase) → ARNtCys-Cystéine
Codon sur ARNm UUG.
ARNtAla + ATP + Alanine + Enzyme spécifique (Alanyl ARNt synthétase) → ARNtAla-Alanine
Codon sur ARNm CCG ou UCG.
In vitro :
- Ribosome + ARNpoly UUG artificiel + ARNtCys-Cystéine + ARNtAla-Alanine → Protéine polyCystéine.
- Ribosome + ARNpoly CCG ou UCG artificiel + ARNtCys-Cystéine + ARNtAla-Alanine → Protéine polyAlanine.
Il est possible de modifier :

Il n’y a pas de rapport direct Codon-acide aminé. L’affinité se fait Codon-ARNt. Existence d’un ARNt et de sa spécificité : adaptateur : positionne les acides aminés sur l’ARNm.
III.C.3. Structure des ARN transfert

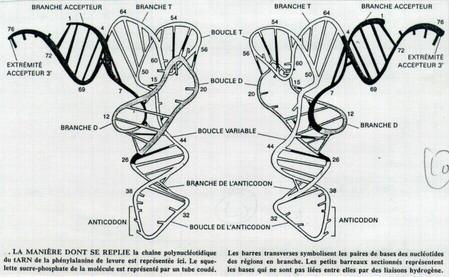
Structure en trèfle. 4 bases + bases mineures : motif de base majeur après transcription.
Boucles : séquences complémentaires en disposition inverse.
Sur 3’ se fixent les acides aminés. 3’ = ACC.
Boucle supplémentaire variable. Dans l’espace, se spiralise en hélice.
Anticodon : 2 nucléotides bien orientés + 1 mal orienté.
III.C.4. Biogenèse des ARNt
A partir des gènes des ARNt → fabrication sous forme de pré ARN (formes plus longues), puis phénomènes d’excision : clivages. Certains peuvent être fabriqués par 2.

IV. Le code génétique
Colinéarité gène-protéine → séquençage des protéines, séquençage de l’ADN → correspondance 1980. Découverte en 1960.
IV.A. Le code génétique est à « 3 lettres »
IV.A.1. Hypothèse d’un code à 3 lettres
Coder pour 20 acides aminés différents avec 4 nucléotides différents.
1 nucléotide pour 1 acide aminé → 4 acides aminés
2 nucléotides pour 1 acide aminé → 16 acides aminés
3 nucléotides pour 1 acide aminé → 64 acides aminés
Rejet de l’hypothèse 4 nucléotides pour 1 acide aminé : trop important/nécessité. Stock plus important inutile (loi du moindre effort).
IV.A.2. Preuve d’un code à 3 lettres
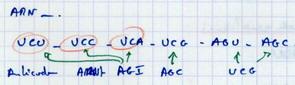
Preuves expérimentales. Quand on a su fabriquer des ARNm de synthèse, grâce à la polynucléotide phosphorylase qui hydrolyse les ARN en ribonucléotides (mais réaction légèrement réversible) : |
 |
Ne nécessite pas de matrice, mais nécessite une amorce. Fabrication de co-polymères mixtes. 2 fois plus d’ADP pour 1 GDP dans le tube à essais (2/3 A et 1/3 G). |
|
IV.A.3. Le code génétique
Crick avait remarqué des mutations avec délétions ou additions de 1 ou 2 nucléotides étaient catastrophiques alors qu’avec 3 nucléotides, ces mutations étaient minimes.

IV.B. Les caractéristiques du code génétique
IV.B.1. Il n’est pas chevauchant
1 nucléotide coderait successivement pour 3 acides aminés s’il était chevauchant (lysine, arginine, acide glutamique). |
IV.B.2. Le code génétique est dégénéré ou redondant
On s’attendait à trouver 20 codons. Or il y en a 61 et 3 non-sens : Ocre, Ambre et Azur. Donc, plusieurs codons codent pour le même acide aminé (exemple : 6 codons différents codent pour la leucine). Ceci présente un intérêt évident pour les mutations. Si 20 codons et 44 non-sens alors une mutation entraînerait dans 2/3 des cas un codon non-sens et donc l’arrêt de la synthèse protéique. Mais le code est redondant, donc une mutation ponctuelle peut ne pas entraîner de mutation. Si l’acide aminé est différent, la protéine se fera quand même. Peut-être moins efficace, mais elle existera (exemple de l’anémie falciforme). Cela évite que les mutations soient létales.
IV.B.3. Oscillations du 3ème nucléotide = Wobble

I = Inosine = adénine désaminée (base mineure).
Modification de l’adénine après transcription.
Manque de rigueur. Le 3ème nucléotide a moins d’importance que les 2 premiers.
XY|A XY|U
|G |C
10 codages à 2 lettres ® 16 acides aminés différents.
Variations au niveau de l’expression, mais pas de l’information génétique. Conduit au même acide aminé. Pas d’étroite spécificité, mais correspondance au même acide aminé.
IV.B.4. Le code génétique est universel

Les ARNt spécifient les mêmes acides aminés chez les procaryotes et les eucaryotes. Mêmes codons → mêmes acides aminés.
Exceptions :
- Certaines bactéries et mitochondries des mammifères : UGA normalement non-sens code pour le tryptophane et AGA et AGG non-sens.
- Chez les protozoaires ciliés seul Azur est un non-sens.
Ces variations de détail sont très peu généralisées. Elles correspondent à des mutations. Le code génétique reste universel. Intérêt évolutif : la vie a une origine unique.
V. Mécanisme de la synthèse des protéines chez les procaryotes
![]()
V.A. La transcription du code génétique
Formation d’ARN au contact de l’ADN. L’ADN sert de matrice, et l’enzyme est l’ARN polymérase.
V.A.1. L’ADN sert de matrice : une seule chaîne
Gène = portion d’ADN. Si les 2 brins servent de matrice, alors on obtient 2 protéines différentes. Or 1 gène → 1 protéine. Travaux sur les virus SP8 sur bacille subtil. SP8 possède 2 chaînes d’ADN qui ont des compositions en nucléotides très différentes. La distinction est donc facile. |

V.A.2. Intervention d’ARN polymérase ADN dépendante
Découverte en 1959. Permet la synthèse d’ARN à partir de ribonucléotides tri Phosphates = NTP. Nécessite une matrice mais pas d’amorce. Elongation dans le sens 5’ → 3’. Intervient en présence de magnésium. Exemple : celle du colibacille : σ = rôle régulateur. |
 |
V.A.3. Mécanismes de la transcription
V.A.3.a. Structure d’un gène

V.A.3.b. Initialisation de la transcription
L’ARN polymérase se fixe sur le site de reconnaissance si le facteur sigma est fixé sur le core de l’enzyme. Il peut y avoir en plus des protéines régulatrices. Cette enzyme va glisser au site d’attachement et provoquer l’ouverture de la chaîne d’ADN (séparation des 2 chaînes = boite TATA [CºG T=A]) Elle se fixe sur la partie intérieure et glisse jusqu’au site de début de transcription T. Le premier nucléotide positionné = U. La transcription débute et progresse dans le sens 3’ → 5’.

Très rapidement, le facteur sigma quitte le core de l’enzyme. Son rôle = permettre la fixation de l’enzyme sur la matrice. S’il persiste, il ralentit la transcription.
V.A.3.c. Mécanismes d’élongation
Identique à celui de l’ADN

Progression antiparallèle par le jeu de la complémentarité des bases. L’ARN polymérase est ADN dépendante. La synthèse est rapide : 2 000 paires de bases par minute. Dès q’une dizaine de nucléotides sont assemblés, l’ARN se détache et l’ADN se reconstitue.

L’association ADN/ARN est moins stable que l’association ADN/ADN. Le détachement est spontané. La boucle évolue régulièrement avec un désenroulement vers l’avant qui entraîne un réenroulement vers l’arrière. Plusieurs ARNm peuvent être transcrits l’un derrière l’autre.

V.A.3.d. Arrêt de la synthèse : terminaison

Les bases complémentaires antiparallèles forment une structure en épingle à cheveux. L’ARN polymérase se détache sous l’action du facteur rho. La transcription est terminée. Les signaux de la terminaison (3) :
- Formation de l’épingle à cheveux |
- Succession de nucléotides Adénine | → Fixation du facteur Rho
- Facteur Rho
Il se peut qu’il existe d’autres facteurs de régulation.

La partie leader est nécessaire à la fixation de l’ARNm sur le ribosome. L’épingle à cheveux donne le signal de la fin de la transcription. La queue poly U protège l’ARNm contre des dégradations précoces. Tous les ARNm commencent par le même codon AUG.
L’ARN polymérase largue le facteur Rhô, récupère le facteur Sigma, et recommence. Il existe des inhibiteurs qui se lient à la matrice (Actinomicine D = antibiotique qui bloque la transcription et la réplication) ou à l’ARN polymérase (bloquent la transcription : la rifamycine, seulement chez les procaryotes).

V.B. La traduction
Enchaînement des acides aminés sur les ribosomes selon l’ordre dicté par l’ARNm par l’intermédiaire des ARNt.
V.B.1. Fixation des acides aminés sur les ARNt
20 acides aminés différents – 60 ARNt. Grâce à une enzyme, l’Aminoacyl ARNt synthétase. C’est une enzyme de taille importante.
|
 |
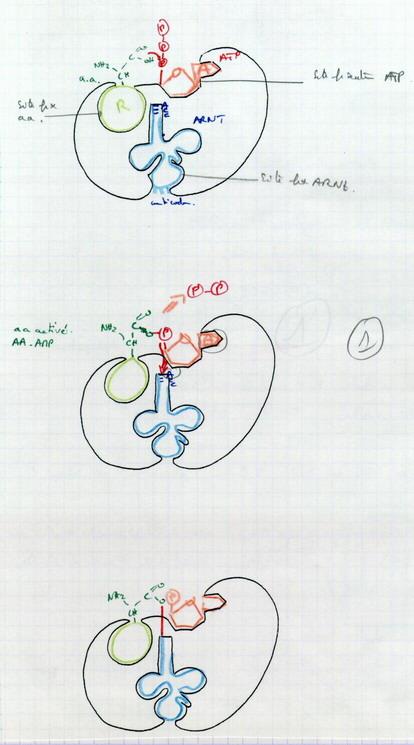
V.B.2. Les étapes de la traduction
V.B.2.a. L’initiation

La synthèse est possible si et seulement si le facteur d’initiation F est associé à GTP.
- Fixation ARNt sur le codon
- Fixation petite Sous Unité sur le complexe ARNm-ARNt
- La grosse Sous Unité vient se mettre en place
- libération du facteur F et hydrolyse du GTP
Sur la grosse SU, 2 sites : le site P « Peptidyp » et le site A « Aminoacyl ».
- Un autre ARNt est fixé. Le facteur d’élongation T est libéré, le GTP est hydrolysé.
- Deux acides aminés cote à cote grâce aux ARNt. Ils se présentent avec le radical amine de l’un au contact de la fonction acide de l’autre.

Le 1er ARNt est libéré.
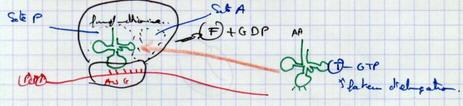
V.B.2.b. Elongation
- Translocation du ribosome

- Fixation nouvel ARNt-acide aminé
- Formation de la liaison peptidique
Translocation du ribosome : transition allostérique provoquée par GTP. Le ribosome possède un demi tunnel → synthèse.
V.B.2.c. Terminaison
Il faut que sur l’ARNm se trouve un codon non-sens : pas de nouvel acide aminé positionné. Intervention d’un facteur de relaxation R-GTP qui va provoquer la dissociation du ribosome. L’ARNm est libéré de ce ribosome. Or le ribosome est associé au polysome. Chaque ribosome synthétise une protéine. |
 |
V.B.3. Bilan énergétique de la traduction
C’est l’énergie dépensée par la cellule pour créer une liaison peptidique.
- 2 liaisons pour activer l’acide aminé (2 x 30 kJ).
- 1 liaison pour fixer l’ARNt sur le codon (30 kJ)
- 1 liaison pour faire la translocation du ribosome (30 kJ)
soit 120 kJ / liaison peptidique créée.
ΔG’0 liaison peptidique ≈ 20 kJmol-1. Ce n’est pas très performant. La création d’une liaison peptidique est un phénomène complexe. Des protéines régulatrices interviennent et le procédé est coûteux en énergie. Mais cela assure la fiabilité du phénomène : sûreté et précision.
V.C. Caractéristiques particulières aux procaryotes
Sur un même gène plusieurs ARNm sont fabriqués les uns derrière les autres. Un gène donne donc de nombreuses molécules d’ARNm. Il y a amplification du phénomène. La traduction représente un deuxième niveau d’amplification. Les phénomènes de transcription et de traduction sont quasi simultanés. Le couplage de ces 2 phénomènes assure l’apparition rapide de protéines. |
 |
VI. Régulation de l’activité des gènes chez les procaryotes
Les protéines cytoplasmiques ne sont pas présentes au même taux dans les cellules. Elles sont synthétisées à des vitesses différentes, et il existe des systèmes régulateurs. Dans la cellule, il existe 2 sortes de gènes :
- Non régulés qui s’expriment continuellement (protéines constitutives).
- Régulés qui ne fabriquent des protéines qu’en fonction des besoins.
- Par induction
- Par répression
- Par induction
VI.A. Régulation par induction
VI.A.1. Mise en évidence
E Coli se développe sur eau + Sels minéraux + Glucose (source à la fois de Carbone et d’Energie). Dans un milieu contenant à la fois du glucose et du lactose, E Coli utilise le glucose, mais pas le lactose. Une enzyme intervient pour la dégradation du glucose. Aucune enzyme n’intervient pour dégrader le lactose. Mais quand on place E Coli sur un milieu composé d’eau, de sels minéraux et de lactose, le colibacille utilise le lactose comme source à la fois de Carbone et d’Energie. On voit apparaître dans la cellule 3 nouvelles enzymes :
- β galactosidase (Lactose → glucose + galactose).
- β galactoside permease qui facilite la pénétration de cette molécule dans la cellule.
- β thiogalactosyl transférase.
Ces 3 enzymes n’existaient pas précédemment. Ce sont des enzymes inductibles qui n’apparaissent que lorsque les besoins se font sentir. Ces 3 enzymes sont fabriquées simultanément : régulation coordonnée. Les études de Jacobs et Monnau montrent que ces 3 enzymes étaient issues d’un gène unique avec formation d’un seul ARNm = ARN polycistronique (cistron = gène). L’ensemble de ce gène a été appelé l’opéron lactose.
VI.A.2. Mécanismes de cette régulation
Problème : comment le lactose induit-il la synthèse coordonnée de ces 3 enzymes ?
VI.A.2.a. Dans le cas normal l’opéron lactose est réprimé
- Observations
Il existe des mutants qui fabriquent continuellement ces 3 enzymes même en l’absence de lactose : ce sont des mutants constitutifs.
- Hypothèse
Dans le cas normal, il existe un gène qui fabrique un inhibiteur protéique et cet inhibiteur vient bloquer l’opéron lactose. Les mutants possèdent un gène de l’inhibiteur non fonctionnel : I-.
- Vérification expérimentale
En créant des diploïdes partiels : dans les cellules I-, on injecte de l’ADN de I+. Dans ce cas, les cellules I- deviennent I+.
- Modèle (provisoire)

Comment cette protéine bloque-t-elle l’opéron lactose ?
VI.A.2.b. La protéine inhibitrice se fixe sur une portion d’ADN différente du gène
- Observations
Ils ont trouvé des souches constitutives qui ne redevenaient pas normales quand on formait un diploïde partiel.
- Hypothèse
L’inhibiteur devait se fixer en un point spécifique de l’ADN : gène opérateur, qui, chez les mutants ne peut fixer la protéine inhibitrice. Nouvelle souche de mutants I+O-.
- Modèle provisoire proposé


VI.A.2.c. Modèle de l’opéron lactose Jacob et Monod
Régulation par induction : le lactose est donc l’inducteur des enzymes qui vont permettre sa propre utilisation.
VI.A.2.d. Confirmation du modèle 1967
- Isolement du répresseur Gilbert 1967
Il recueille des protéines des E Coli. Certaines, réinjectées à la souche I- transformaient I- en I+, cessant d’être constitutives. Cette protéine était donc le répresseur. Cette protéine possédait la propriété de fixer le lactose.
- Cette protéine répresseur se fixe sur l’ADN d’E Coli
Culture sur milieu radioactif → protéines radioactives → donc répresseur radioactif. In vitro, on met en présence le répresseur* et l’ADN du colibacille. On montre que cette protéine se fixe sur l’ADN. Si on ajoute du lactose, cette protéine* ne se fixe pas sur l’ADN. Si l’ADN provenait de mutants O-, le répresseur ne se fixe pas non plus.
VI.A.2.e. Mécanisme d’action du répresseur

VI.A.3. Pourquoi l’induction de l’opéron lactose ne se fait-elle pas en présence de lactose et de glucose ?
VI.A.3.a. Observation
E Coli + glucose + lactose → Utilise le glucose, n’utilise pas le lactose → pas d’induction de l’enzyme d’utilisation du lactose. L’induction ne résulte pas de la seule présence du lactose.
VI.A.3.b. Rôle de l’AMP cyclique
Dans le colibacille, on a observé un taux d’AMPc faible en présence de glucose, et un taux d’AMPc élevé en l’absence de glucose.
En injectant de l’AMPc + lactose + glucose → l’induction des enzymes du lactose se produit. L’AMPc est nécessaire au phénomène d’induction et ce taux n’augmente qu’en l’absence de glucose. Cela explique que le taux soit bas en présence de glucose + fructose.
VI.A.3.c. Intervention de la protéine activatrice du catabolisme ou π CAP
In vitro, lorsque on place de l’ADN + ribonucléotides + ARN polymérase, les gènes de l’opéron lactose sont transcrits très lentement (il n’y a pourtant pas de répresseur).
Si on ajoute de l’AMPc, on n’obtient rien de plus.
Si on ajoute la π CAP, la synthèse n’est pas plus rapide.
Si on ajoute les 2, la synthèse est très fortement stimulée. Ces 2 facteurs agissent sur la 1ère partie du promoteur pour stimuler. La π CAP existe dans la cellule, mais elle est inactive. Elle ne devient active qu’en présence d’AMPc (absence de glucose). Cette protéine va agir sur la partie proximale du promoteur (début) pour faciliter la reconnaissance par l’ARN polymérase. La régulation de l’opéron lactose est sous l’influence de 2 types de contrôles :
- Frein : le répresseur est neutralisé par le lactose
- Accélérateur : les protéines activatrices du catabolisme sont activées par l’AMPc (absence de glucose)
VI.B. Régulations par répression
La présence d’un substrat bloque la synthèse d’enzymes.
VI.B.1. Mise en évidence
E Coli, cultivée sur milieu : eau, glucose, sels minéraux dont sels d’ammonium = source minérale d’azote nécessaire à la synthèse de tous les acides aminés. E Coli possède toutes les enzymes nécessaires à ces synthèses. Régulation par répression : la présence du substrat réprime les gènes responsables de la synthèse des enzymes. |
VI.B.2. Modèle de régulation
Les enzymes sont regroupées en un opéron qui est sous le contrôle d’un seul promoteur et soumis à des gènes régulateurs.

En présence d'histidine:

En fait, il s’agit d’un complexe ARNtHist-Histidine.
Chez les procaryotes, la régulation est coordonnée par des enzymes d’une même voie métabolique. En effet, les gènes de structure sont regroupés en opérons : 1 même promoteur unique et sous contrôle de gènes régulateurs et d’un opérateur.
Cas général : les régulations
- Par induction se font pour les biosynthèses d’enzymes du catabolisme
- Par répression se font pour les biosynthèses d’enzymes de l’anabolisme
Ces régulations s’effectuent dès la 1ère étape : c’est la transcription qui est bloquée → évite la consommation d’ARNm et de protéines = économie de substrat et d’énergie.
Il semblerait qu’il y ait un deuxième niveau de régulation. Les enzymes ne sont pas présentes au même taux dans la cellule. Régulation lors de la traduction → lors de l’expression, les ribosomes ajustent les besoins.
Sujets
Gènes ? caractère, protéine, discussion :
1 gène des protéines
1 gène pas de protéines → opérateurs, ARN ribosomaux ou de transfert
Gènes opérateurs ont un rôle fondamental mais ne sont ni transcrits ni traduits.
