Introduction
Etude sur les organismes eucaryotes haploïdes. Le phénotype est le reflet direct du génotype : pas de problèmes de dominance, codominance...
Champignons filamenteux et quelques algues.
I. Transmission d'un couple d'allèles
Le méiose est à l'origine de spores situés dans une enveloppe (asque). Ces spores sont appelés ascospores.

I.A. Cas des tétrades non ordonnées
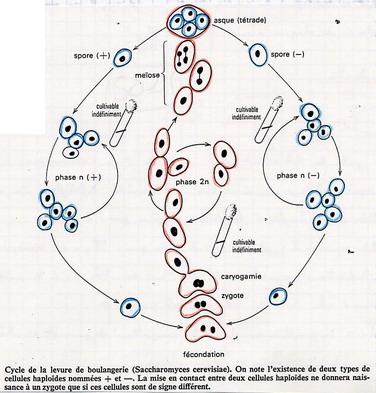
I.A.1. Cycle Haplodiplophasique
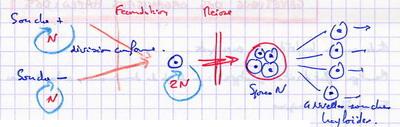
I.A.2. Exemple caractère : aptitude à synthétiser le tryptophane

Culture de spores pour déterminer le génotype. Seules les souches Trypt+ se développent (1 sur 2 par asque). |
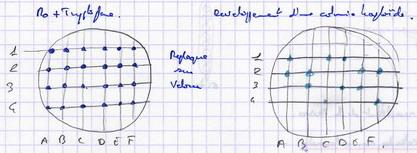 |
Généralisation :
 |
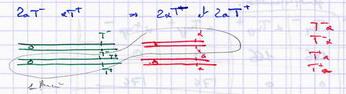
Dans le zygote les 2 allèles sont présents même si le - ne s'exprime pas. Au départ, 2 allèles impliqués a et a'. A l'arrivée, deux fois plus : 2a et 2a'. Les allèles ont été dupliqués pendant la méiose. Les allèles réunis dans le zygote sont séparés dans les spores : il y a eu ségrégation des allèles. |
Duplication et ségrégation or le comportement des allèles lors de la méiose est identique à celui des chromosomes (les gènes sont portés par les chromosomes). On observe un parallelisme étroit entre le comportement des gènes et des allèles : 2 chromatide, puis séparation des chromosomes homologues.
I.B. Cas des asques ordonnés
Les spores sont alignés. C'est le cas chez de nombreux champignons filamenteux. Ex : Neurospora crassa et Sordaria.
|
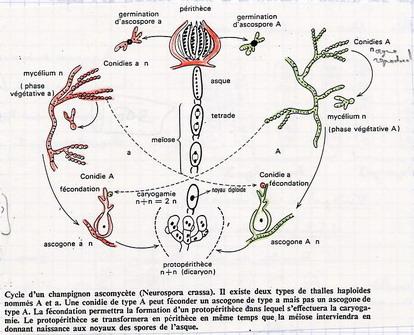 |
I.B.1. Le cycle des champignons filamenteux
Conidie = organe reproducteur filamenteux qui libère des cellules (multiplication végétative).
Ascogone =
organe reproducteur de la reproduction sexuée.
Lorsque ces organes apparaîssent, la fécondation croisée est poosible entre les souches A et a. Chaque souche est à la fois male et femelle. Il y a fusion des deux cellules, mais pas des noyaux : cellule diploIde avec deux noyaux non fusionés = dicaryon.
Ces cellules se divisent et donnent des filaments dicaryotiques (protopérithèce) = préfructification. Dans les cellules situées aux extrémités, les noyaix fusionent (caryogamie). L'originalité provient du décalage ente la fusion des cellules et la fusion des noyaux. Une fois les noyaux fusionnés, il y a méiose suivie de mitose pour donner 8 cellules. Les fuseaux sont parallèles, ce qui donne une file de cellules.

I.B.2. Etude d'un exemple
Etude d'un caractère : couleur de la spore, directement observable dans l'asque.
Deux allèles : blanc / noir → directement observable
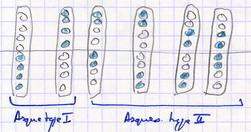 |
p = fréquence des allèles de type II. Chaque asque de type II a une fréquence p/4. 1 - p = fréquence des asques de type I. Chaque asque de type I a une fréquence (1 - p)/2 |
I.B.3. Interprétation des observations
I.B.3.a. Interprétation des observations
- Formation des asques de type I
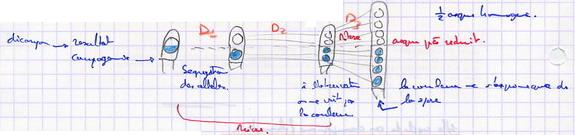
La ségrégation des allèles s'opère dès la première division. On dit qu'il y a préréduction.
- Formation des asques de type II (½ asques non homogènes)
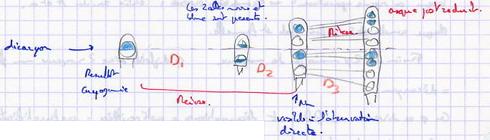
La ségrégation définitive se fait lors de la seconde division. On dit qu'il y postréduction. Dans tous les cas, D3 est une division conforme (mitose).
D1 et D2 constituent quelque chose de particulier (méisoses).
I.B.3.b. Interprétation chromosomique
- Cas de préréduction
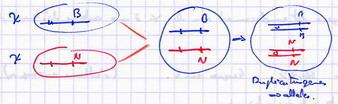
- Sans crossing over

- Avec crossing over
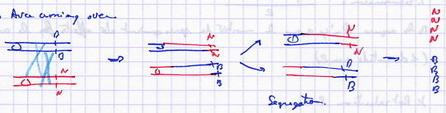
- Cas de postréduction (asques de type II)

I.B.4. Pourcentage de postréduction et signification
I.B.4.a. Définition
(Nombre d'asques postR / Nombre d'asques) x 100
I.B.4.b. Signification
Ce pourcentage est constant pour un couple d'allèles donné. Il varie d'un couple à un autre. En supposant que tous les crossing over soient équiprobables entre tous les points d'un chromosome, on peut supposer que le crossing over sera pour un allèle donné d'autant plus probable que l'allèle est éloigné d'un centromère. Si le porcentage de postR est constant pour un couple donné, alors l'allèle est toujours à la même distance du centromère.
On a appelé locus du gène cet emplacement sur le cr*hromosome, à une certaine distance du chromosome. Ce pourcentage ne peut pas dépasser 66,67 % (4 asques sur 6) et s'adresse aux asques.
I.B.5. Pourcentage de recombinaison
I.B.5.a. Définition
Pourcentage qui s'adresse au pourcentage de recombinaison d'allèles = crossing over : pourcentage de spores résultant d'un crossing over.
% Recombinaison = (% postRéduction ) / 2
Dans les asques, il n'y a que la moitié des spores qui ont été affactés par le crossing over (1 chromatide sur 2).
% postréduction = P
% préréduction = 1 - P
Nombre de spores résultant d'un crossing over = 4 P (8 spores par asque)
Nombre total de spores = 8 (P + (1 - P))
% spores recombinés = 4P/8 = P/2
I.B.5.b. Intérêt
Faire une estimation de la distance entre le locus du gène et son centromère (on suppose l'équiprobabilité des crossing over). Plus le pourcentage de recombinaison sera élevé, plus il sera loin du centromère. Plus le gène est proche du centromère, plus le pourcentage de recombinaison sera faible.
UG = Unité Génétique = centimorgan
| 1 Cm = 1 UG = 1 % recombinaison génétique %postR = 48 % % Recombinaison = 24 % soit 24 % de crossing over donc le locus est situé à 24 UG du centromère. |
I.B.5.c. Précision - Estimations
Tant que le résultat trouvé reste inférieur à 15 ou 20 Cm, la mesure est bonne. Quand le résultat est supérieur, la mesure est sous estimée (2 crossing over peuvent s'annuler).
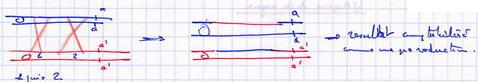
Les 2 crossing over vont passer inaperçus et donc la valeur sera sous estimée.
II. Transmission de deux couples d'allèles
Deux cas :
- Les allèles sont indépendants (sur deux chromosomes différents)
- Les allèles sont liés (portés par le même chromosome)
II.A. Ségrégation indépendante des allèles
Exemple : Neurospora.
Deux caractère : Signe sexuel (2 allèles a et α) et Auxotrophie par rapport au Tryptophane (2 allèles Tryp+ et Tryp-).
Croisement : aTryp- x
αTryp+
II.A.1. Analyse des spores
| 25 % de spores aTryp- | Génotypes parentaux |
| 25 % de spores αTryp+ | |
| 25 % de spores aTryp+ | Génotypes recombinés |
| 25 % de spores αTryp- |
On assiste à une recombinaison des gènes et à l'apparition de deux nouveaux phénotypes (ségrégation des allèles).
II.A.2. Analyse des asques
II.A.2.a. Les différents types
- Asques où l'on trouve uniquement des spores aTryp- et αTryp+ = génotypes parents. Ce sont les asques ditype parentaux = DP.
- Asques à deux types aTryp+ et αTryp- = génotypes recombinés = ditypes recombinés = DR.
- Asques à 4 types de spores aTryp+, aTryp-, αTryp+ et αTryp- = génotypes parentaux et recombinés = tétratype = T.
En fréquences, on trouve
6 types d'asques DR (C42)
6 types d'asques DP (C42)
24 types d'asques T (4!)
soit un total de 36 types d'asques différents.
II.A.2.b. Origine de ces asques

II.A.2.c. Fréquence
DR = 87 asques
DP = 83 asques
T = 135 asques
DR ≈ DP
T > DP et DR
II.A.3. Interprétation de ces observations
| Autant de spores recombinées que de génotypes parentaux Parental : aTryp- et αTryp+ Recombiné : aTryp+ et αTryp- |
 |
Les allèles simultanément présents chez les parents n'ont aucune tendance particulière à rester liés : ils peuvent se recombiner facilement : ils ne sont pas liés, mais indépendants. La recombinaison est facile et aléatoire.
4 x 25 % et DP = DR → les gènes sont indépendants et portés par des chromosomes différents.
Il y a parallélisme entre le comportement des deux gènes qui ségrèguent indépendamment l'un de l'autre et celui des paires de chromosomes homologues lors de la méiose lorsque l'on constate qu'une chromatine d'une paire a autant de chances de se retrouver avec un chromatine de la deuxième paire. Analogie de comportement
→ les allèles sont portés par des chromosomes différents.
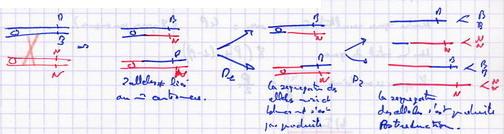
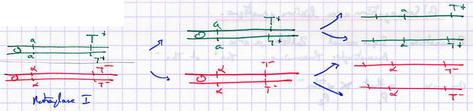
- Formation des DP
- Par préréduction sans crossing over
- Par postréduction avec crossing over

- Formation des DR
- Par préréduction sans crossing over
- Par postréduction avec crossing over

- Formation des tétratypes T
- 1 seul crossing over
| Dans les fréquences, T > DP ouDR. Or, il existe des cas particuliers où DP = DR avec T = 0. Il n'y a pas de postréduction : les deux gènes sont très proches du centromère. C'est aussi valable pour les asques ordonnés. Dans le cas des levures, on ne peut pas calculer la distance entre le gène et le centromère, sauf si T = 0 (dans ce cas, le gène est très proche du centromère). |
 |
II.B. Ségrégation avec liaison génique
Deux caractères : Signe sexuel (2 allèles a et α) et Auxotrophie par rapport au Thréonine (2 allèles Thr+ et Thr-).
Croisement : P1 = aThr+ x P2 =
αThr-
II.B.1. Analyse des asques
- Les différents types d'asques
Sur les 210 différents :
136 DP (2 spores athr+ et 2 spores αThr-)
4 DR (2 spores αthr+ et 2 spores aThr-)
70 T (αthr+[R1], athr+ [P1], aThr- [R2], αThr- [P2] - Nombre de spores
| P1 aThr+ → 342 | Génotypes parentaux |
| P2 αThr- → 342 | |
| R1 aThr- → 78 | Génotypes recombinés |
| R2 αThr+ → 8 |
% Recombinaison = (4DR + 2T) / 4(DR + DP + T) x 100 ≈ 18,6 %
II.B.2. Interprétation
- DP > DR
Les gènes qui étaient simultanément présents chez les parents ont tendance à rester liés dans la descendance : ce sont des gènes liés. - Interprétation chromosomique
On peut expliquer que ces deux gènes sont liés si ils sont portés par le même chromosome. On peut expliquer la proportion de DR par les crossing over entre ces deux gènes. 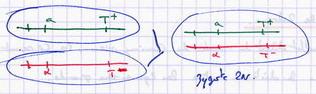
- Formation des DP
- Sans crossing over
- Avec crossing over
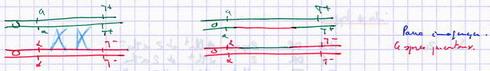
- Formation des DR
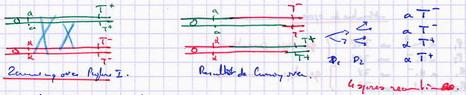
- Formation des T

- Résumé
DP > DR → Gènes liésPas de crossing over → DP
2 crossing over → DR
1 crossong over → Tor les crossing over sont exceptionnels → DP > DR
T > DR
On peut calculer la distance entre les 2 loci.
II.B.3. Distance ente les 2 loci
| Le pourcentage de recombinaison est le reflet direct du nombre de crossing over Les crossing over seront d'autant plus fréquents que la distance entre les deux gènes sera grande. On va utiliser le % de recombinaison pour évaluer la distance entre les 2 gènes. % Recombinaison = 18,6 % |
Remaque : si DP = DR alors les gènes sont indépendants, portés par des chromosomes différents.
Autre cas : gènes liés mais très éloignés l'un de l'autre. Ils peuvent être très souvent séparés et le pourcentage peut tendre vers 50 % (ce qui équivaut au comportement de gènes indépendants : DP se rapproche de DR).
III. Etablissement de la carte génétique
Carte génétique = Carte factorielle
III.A. Le principe
L'objectif est de positionner les gènes les uns par rapport aux autres. On estime la distance entre les gènes et le centromère ou gène - gène si ils sont liés (étude de gènes 2 à deux). Etude des asques.
III.B. Exemple
Chez Neurospora, étude des allèles c/c' (gène de la forme des conidiophores) et p/p' (gène de la couleur des spores).
cp x c'p'
| Asques : 401 DP 2 DR 94 T |
DP > DR → les gènes liés chez les parents ont tendance à rester liés au cours de la méiose (portés par le même chromosome). |
| % Recombinaison entre les 2 gènes = distance entre les 2 gènes = (4DR + 2T) / 4(DP + DR + T) = 9,9 % | 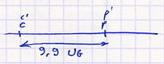 |
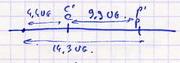
| Couple c/c' % postR = 8,8 % Couple p/p' % postR = 28,6 % % Recombinaison entre centromères et locus gène pour c/c' % Recombinaison = 8,8/2 = 4,4 % pour p/p' % Recombinaison = 28,6/2 = 14,3 % Mathématiquement exact car la distance < 20 UG. |
 |


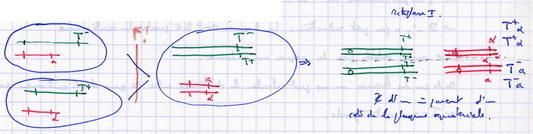
III.C. Le test des 3 points : cas des 3 gènes liés
Valable seulement pour 3 gènes liés a/a', b/b', c/c'. On les considère 2 à 2.
Gènes a et b : a'b' x ab Gènes a et c : ac x a'c' Gènes b et c : bc x b'c' |
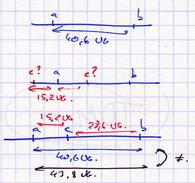 |
L'étude du gène entre les deux a éliminé une partie des doubles crossing over qui s'étaient produits mais qui avaient échappé à l'observation.
La distance entre c et b est aussi sous estimée (> 20).
Pour affiner, il faudrait trouver un autre gène entre b et c. Le 3ème point permet d'affiner la mesure.
Groupe de liaison : regrouper tous les gènes qui sont liés et savoir combien de groupes on obtient. Le nombre de groupes de gènes liés donne le nombre de chromosomes.
iV. Recombinaison intragénique
IV.A. Résultats
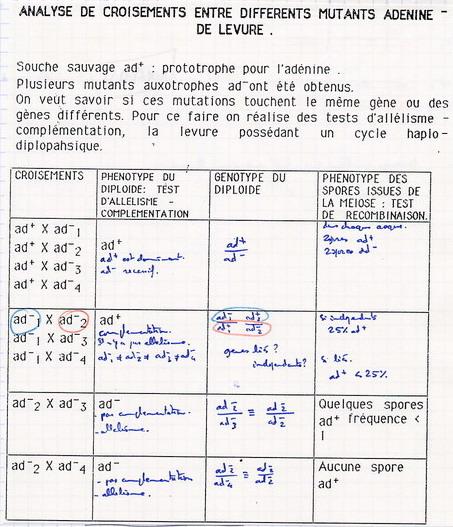
IV.B. Interprétation
- Combien de gènes impliqués ?
4 souches ad-, mais en fait seulement 2 souches : ad1- et ad2- = ad3- = ad4-.
2 gènes seulement : gène 1 et gène 2 = gène 3 = gène 4.
- Cas ad2- x ad3-
S'explique par une recombinaison intragénique. On suppose que ce n'est pas la même partie du gène qui est mutée, et un crossing over entre les 2 points de mutation peut redonner un gène normal.
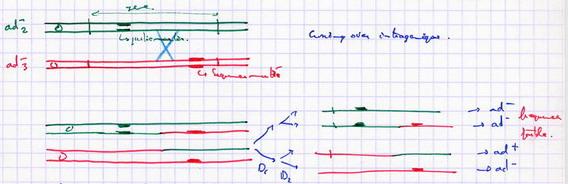
La fréquence est faible car la probabilité de crossing over entre les deux zones mutées est très faible. C'est le même gène qui est muté, mais pas au même endroit : hétéroallèle.
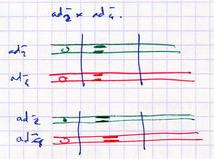 |
Si même mutation alors les crossing over ne peuvent redonner un gène fonctionnel ad+. Si deux mutations légèrement décalées mais chevauchantes, alors les crossing over ne peuvent redonner un gène fonctionnel ad+. |
